Biologie structurale du SARS-CoV-2 : cristallographie aux rayons X
Date de publication: 07-08-2021
Mise à jour le: 16-05-2025
Sujet: Covid-19
Temps de lecture estimé: 1 min
Auteur d'articles
Eufemia PutortiRédacteur médical
Massimo Degano
Rédacteur et traducteur
Viktoryia LuhakovaLa pandémie de COVID-19 en cours a obligé la communauté scientifique à se consacrer à la recherche et au développement de diverses stratégies de lutte contre l'infection par le SARS-CoV-2. La seule façon avisée d'identifier de nouveaux antiviraux est la connaissance de la structure tridimensionnelle, littéralement la forme, des protéines du virus qui sont responsables de l'infection des cellules et de la réplication du virus. Une fois la structure des protéines connue, il est possible pour les chimistes de synthétiser des composés qui bloquent sa fonction, et par conséquent le cycle de vie du virus.
Nous en parlons dans ce cycle de 5 épisodes avec l'aide du Dr Massimo Degano, chef de groupe de l'unité de biocristallographie de l'hôpital San Raffaele et professeur à notre université.
Nous avons expliqué dans les épisodes précédents que, pour développer des médicaments contre le SARS-CoV-2, il est nécessaire de connaître de manière très détaillée (résolution) la structure des protéines du virus que nous avons choisies comme cibles en fonction de leur importance dans le cycle de vie du virus. C'est la tâche de la biologie structurale, et nous avons déjà expliqué le principe de fonctionnement de la cryo-microscopie électronique. La première technique qui a permis de visualiser les structures des molécules avec un détail atomique, et qui fournit encore un détail très élevé, est la cristallographie aux rayons X.
Succès (et contributions) de la cristallographie aux rayons X
La théorie cristallographique a été développée peu après la découverte des rayons X par Wilhelm Röntgen, grâce aux contributions de Max von Laue (lauréat du prix Nobel en 1914) et de William Henry et William Lawrence Bragg (père et fils, tous deux lauréats du prix Nobel en 1915), et au fil des ans, elle a été constamment affinée et améliorée. À l'époque de ces pionniers, déterminer la structure de petites molécules telles que l'aspirine était un formidable défi, mais grâce à des visionnaires tels que Sir John Kendrew et Max Perutz, il a été possible de déterminer les premières structures de protéines, la myoglobine et l'hémoglobine. De 1945 à aujourd'hui, la liste des succès de la cristallographie pour nous fournir des images de protéines d'intérêt biomédical est infinie, et l'importance de ces informations est corroborée par l'Académie royale des sciences de Suède qui récompense régulièrement par le prix Nobel les études structurelles qui nous permettent de comprendre le fonctionnement des protéines complexes (https://www.iucr.org/people/nobel-prize).
Utilisation des rayons X pour visualiser la structure des grandes molécules biologiques
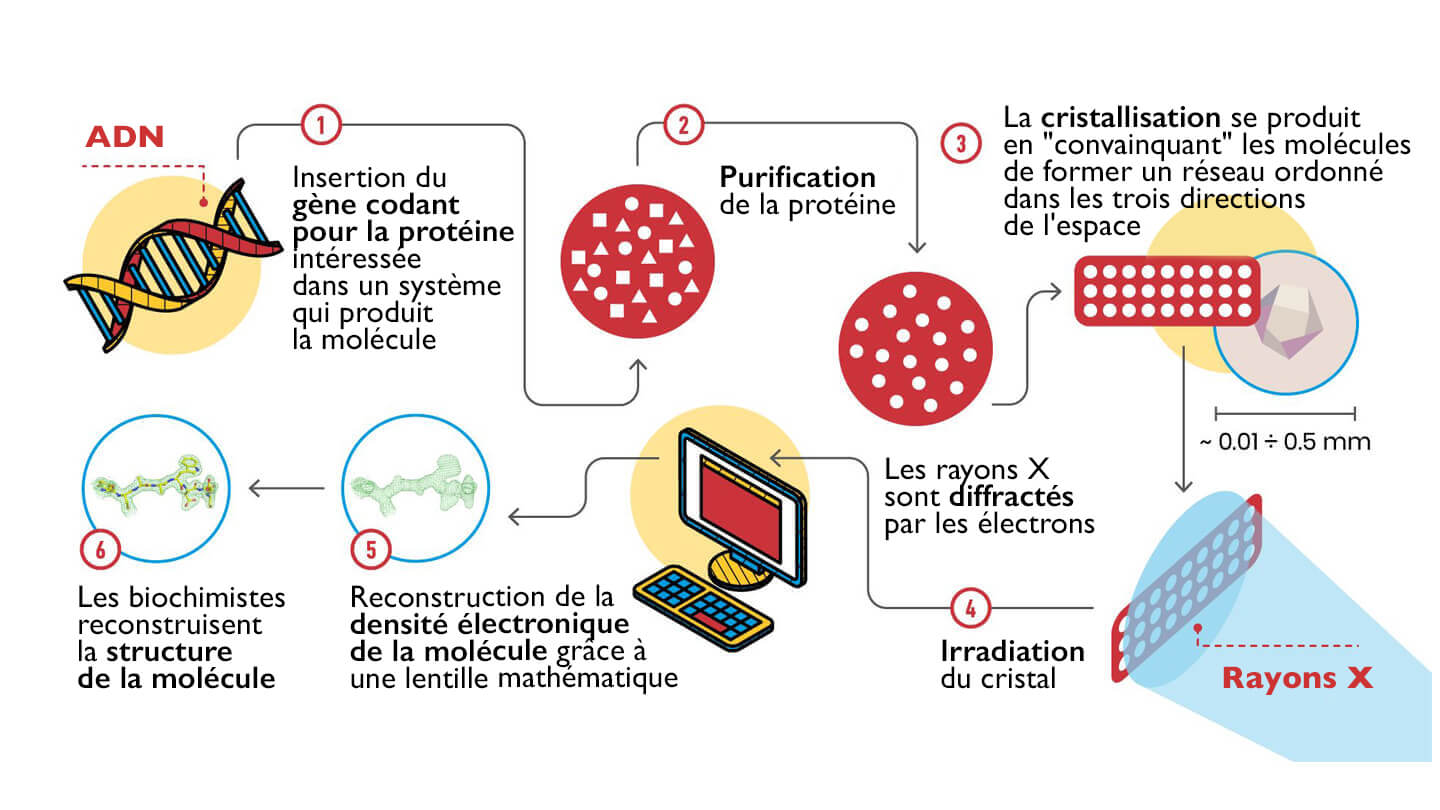
Quelles sont les étapes de cette technique ? Examinons le schéma :
1. 2. Tout d'abord, il est nécessaire de disposer de milligrammes d'échantillon pur, c'est-à-dire de quantités infinitésimales de contaminants : en pratique, de nombreuses molécules identiques.
3. Ensuite, ces molécules doivent former un cristal, c'est-à-dire un réseau tridimensionnel ordonné. Cette étape est fondamentale, car les molécules du cristal sont positionnées selon des motifs de symétrie, tels que ceux étudiés par M. C. Escher (https://mcescher.com/gallery/symmetry/).
4. L'ordre des molécules dans le cristal fait que les électrons autour des atomes, lorsqu'ils sont irradiés par des rayons X, diffusent le rayonnement en phase, s'additionnant comme les composantes d'un chœur vocal. Cet effet d'amplification nous permet de mesurer le spectre de diffraction, c'est-à-dire les positions et l'intensité des rayons X diffractés par les molécules du cristal.
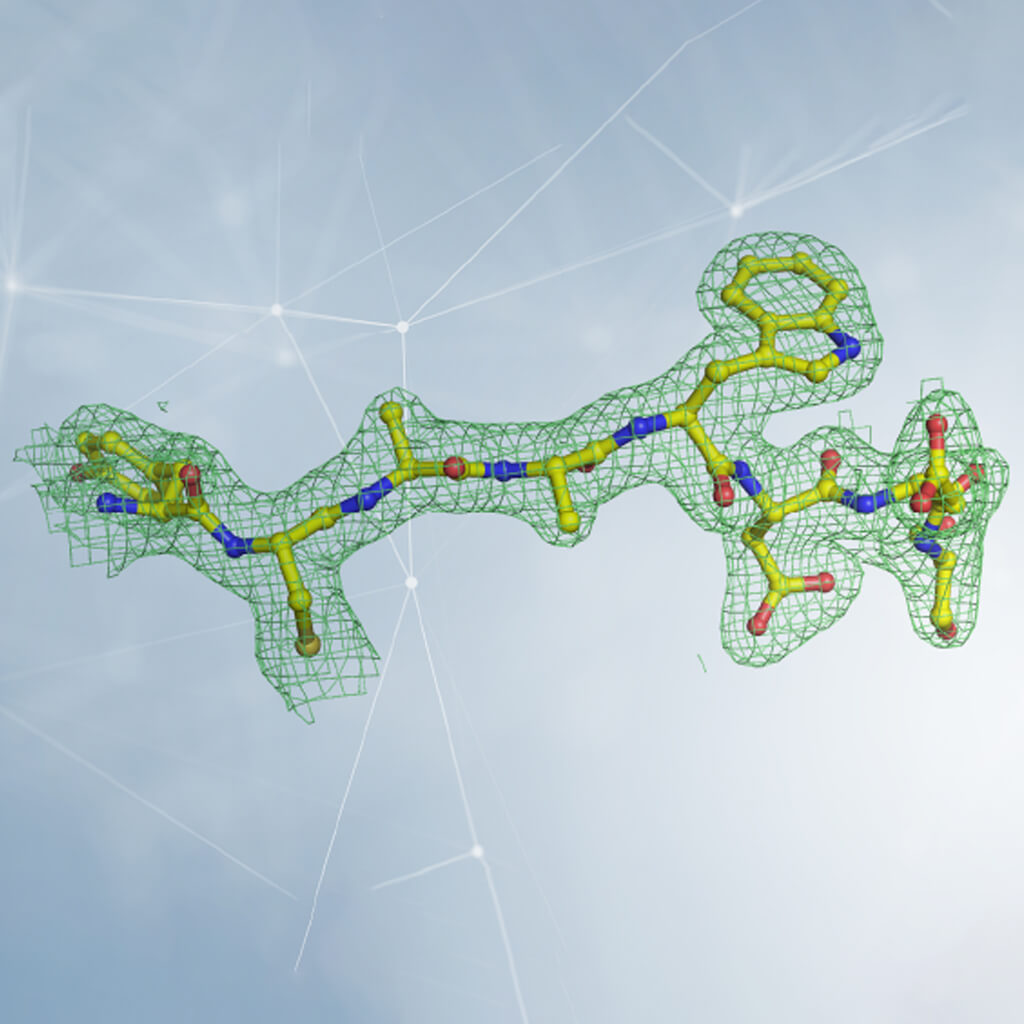
5. Une fois cette mesure effectuée, et en appliquant une sorte de lentille mathématique (appelée "transformée de Fourier inverse"), on obtient la carte de densité électronique qui nous indique comment les atomes sont disposés dans le cristal. La densité électronique peut être visualisée sur l'ordinateur, et les composants de la molécule biologique que nous étudions (acides aminés pour les protéines, bases ribose et azotées pour l'ADN et l'ARN...) doivent y être insérés, dans un emboîtement qui pourrait ressembler à certains jeux vidéo.
6. Ayant terminé l'interprétation de la densité électronique, nous avons enfin la structure tridimensionnelle !
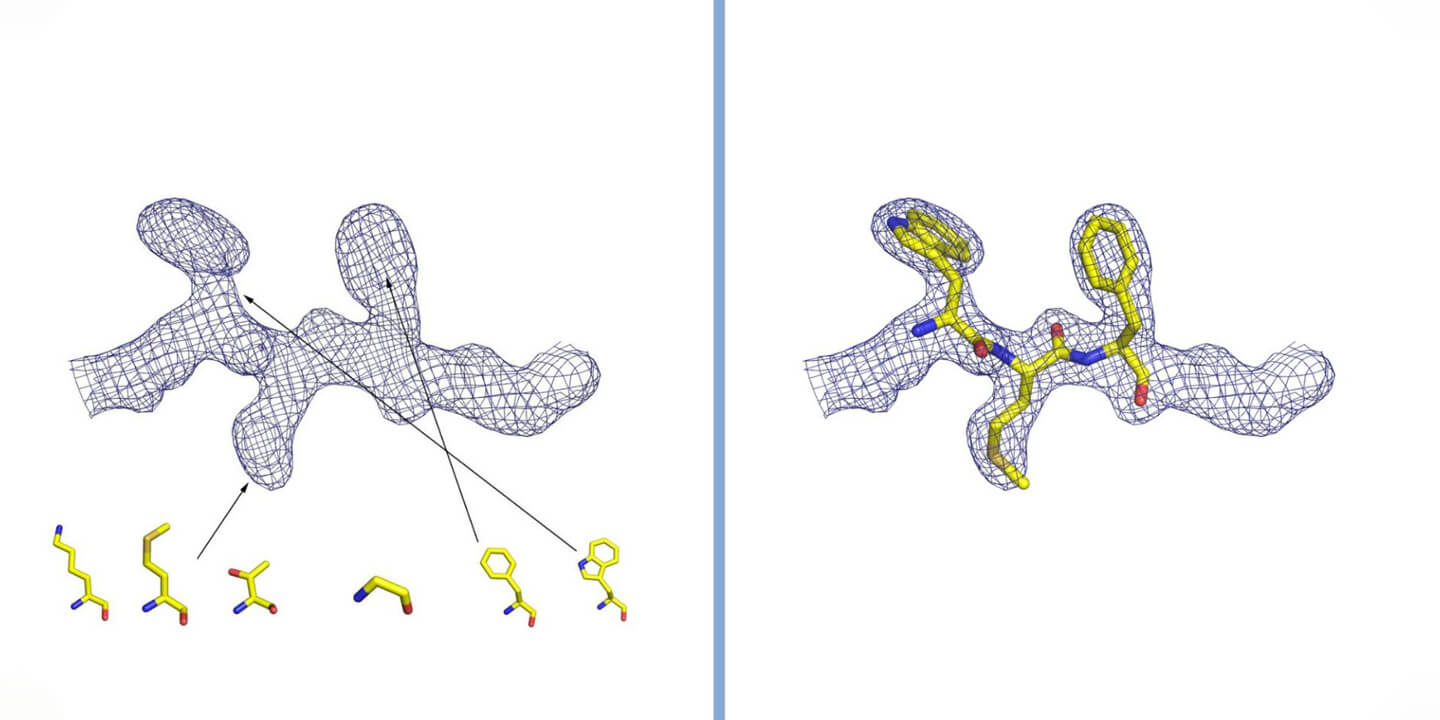
L'interprétation de la densité électronique en cristallographie. La densité est représentée par une grille, à l'intérieur de laquelle se trouvent les atomes qui composent la protéine. Dans le cas d'une protéine, la forme de la densité est observée et comparée à la structure des vingt acides aminés qui la constituent (dont certains sont représentés sur la figure). En insérant les acides aminés dans le bon ordre et en suivant l'enroulement tridimensionnel de la densité électronique, on reconstitue la structure de la protéine. Avec l'aimable autorisation du Dr Degano.
Utilisations de la cristallographie
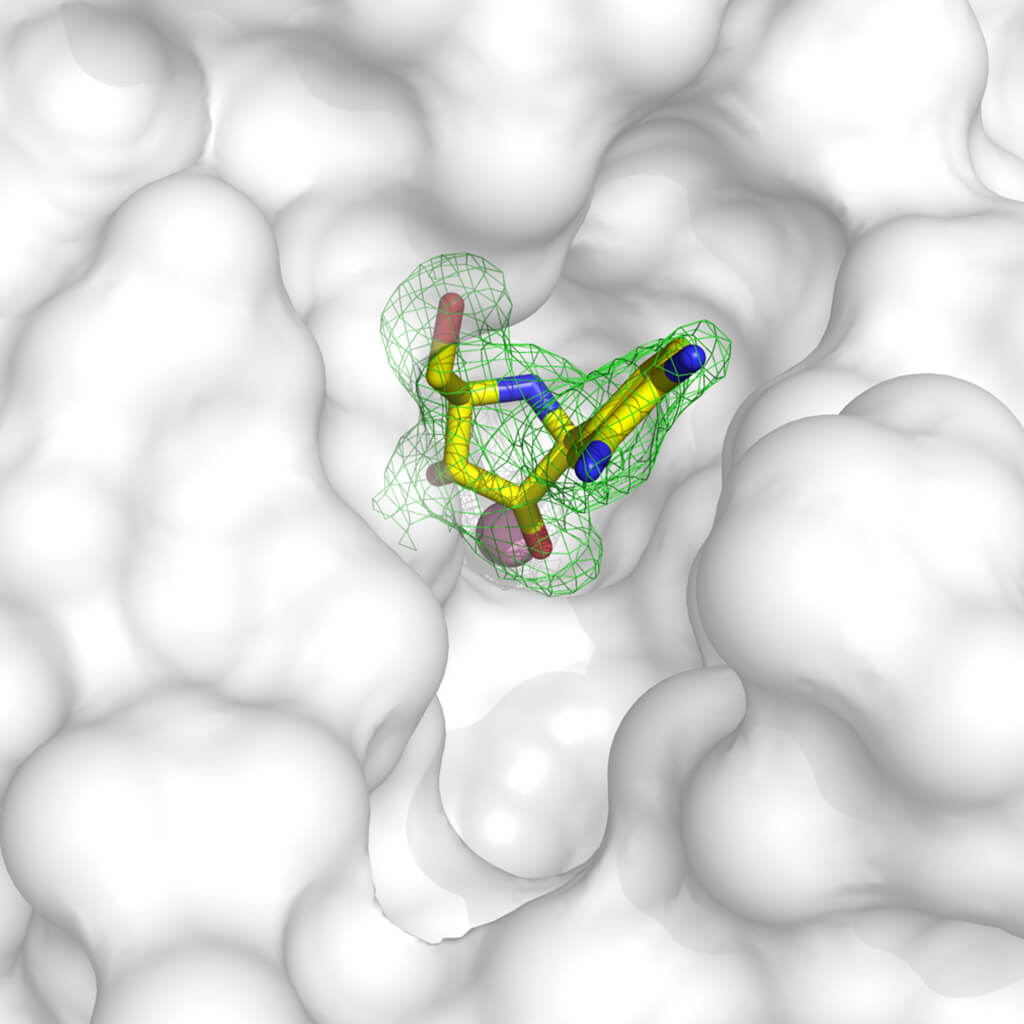
La cristallographie est un outil très polyvalent et puissant pour le développement de composés principaux susceptibles de devenir des médicaments. Grâce à diverses méthodologies, il est possible d'obtenir la structure de protéines cibles liées à des molécules ayant une activité inhibitrice : en effet, il est possible de "tremper" les cristaux dans une solution dans laquelle une substance que nous savons dissoute peut se lier à la protéine cible, ou d'obtenir des cristaux de protéine après l'avoir liée au composé. Une fois que la structure de la protéine liée à un composé est connue, nous pouvons visualiser quelles parties de la protéine (que nous pouvons assimiler à un gant) ne sont pas correctement remplies par la molécule (les doigts d'une main). Grâce à ces informations, nous pouvons modifier la molécule inhibitrice pour qu'elle s'adapte mieux à notre cible, et bloquer sa fonction !
Exemple d'inhibiteur d'une enzyme qui a été inséré dans les molécules d'un cristal. La densité électronique en vert définit clairement où la molécule est positionnée dans la structure de la protéine, représentée par la surface blanche. On peut voir que la molécule n'occupe pas parfaitement la cavité disponible, il est donc possible de concevoir un composé qui complète mieux la structure de la protéine et bloque efficacement sa fonction. La structure utilisée est accessible avec le code 3MKN dans la Protein Data Bank. Avec l'aimable autorisation du Dr Degano.